植生生态所植物逆境中心合作揭示植物SUVH家族组蛋白甲基化酶的调控机理
文章来源: | 发布时间:2018-08-30 | 【打印】 【关闭】
8月28日,PNAS杂志在线发表了中科院分子植物卓越中心/植物生理生态研究所上海植物逆境生物学研究中心杜嘉木研究组和美国加州大学洛杉矶分校Steven Jacobsen研究组合作完成的题为“Mechanistic insights into plant SUVH family H3K9 methyltransferases and their binding to context-biased non-CG DNA methylation”的研究论文。该论文从结构生物学、生物化学、基因组学等层面多角度的对植物特有的SUVH家族组蛋白H3K9甲基化酶开展了研究,发现了这类酶的调控机制,并成功解析了植物CHG位点甲基化序列偏好性的分子基础。
DNA甲基化是一类重要的表观遗传标记,对维持基因组的稳定性有着重要的意义。植物DNA甲基化严格受到组蛋白修饰H3K9me2的调控。植物中H3K9的甲基化主要由植物特有的SUVH家族蛋白来完成,通过其SRA结构域结合甲基化的DNA,并通过其SET结构域来进行组蛋白甲基化。然而这类酶的生化调控机制却研究很少。
本研究中,研究人员首先对SUVH家族成员SUVH6开展了结构生物学研究,测定了SUVH6与甲基化的DNA、辅因子SAM等不同组合的多个状态的复合物的晶体结构。通过结构分析,发现SUVH6的肽底物结合通道受到自身的自抑制调控。并且,SUVH家族的蛋白结合甲基化DNA更依赖于其thumb loop,而不同于哺乳动物的UHRF1更依赖于NKR finger。在此基础上,研究人员测定了SUVH4/5/6三个同源冗余基因和不同序列的甲基化DNA的结合常数,发现植物中负责H3K9甲基化的主效基因SUVH4更加偏好CAG/CTG甲基化,SUVH5更偏好CCG甲基化,SUVH6则没有偏好性,成功的解释了拟南芥中CAG/CTG甲基化明显高于CCG甲基化的原因。合作方的全基因组Chip-seq实验也验证了这个解释。本项研究大大加深了人们对SUVH家族甲基化酶的调控和体内序列偏好性的认识。
植物逆境中心研究生李雪琴是该论文的共同第一作者,杜嘉木研究员和Steven Jacobsen教授是该论文的共同通讯作者。该研究工作受到了科技部国家重点研发计划,基金委,植物分子遗传国家重点实验室,以及中科院的经费资助。
论文链接:https://doi.org/10.1073/pnas.1809841115
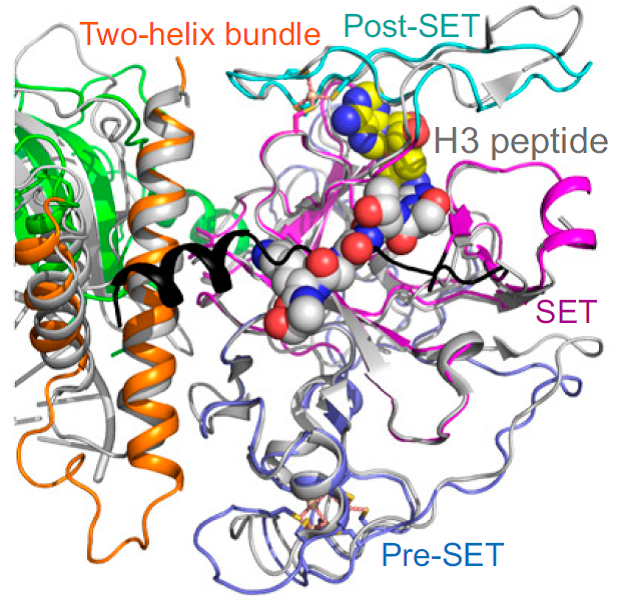
SUVH6受到自抑制调控
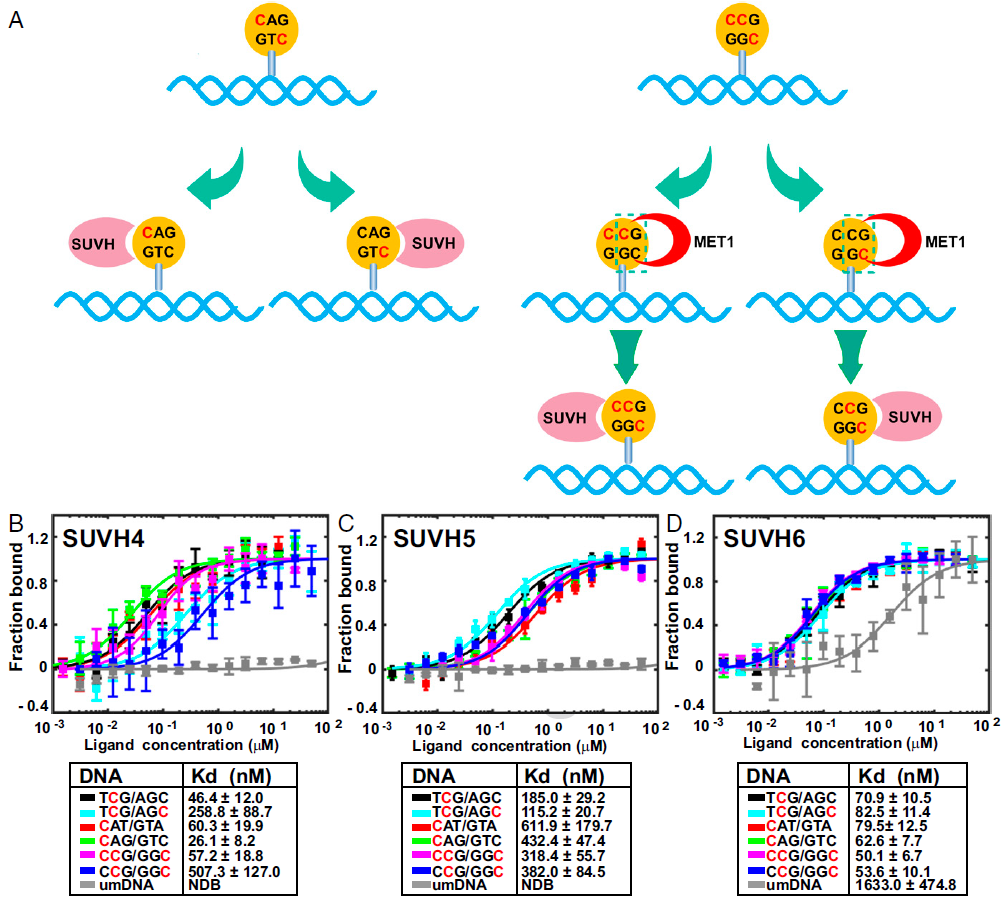
不同SUVH家族成员有不同的序列偏好性
