大数据中心合作发布基于化合物抗纤维化特征的药物重定位平台
文章来源:上海营养与健康研究所 | 发布时间:2020-07-01 | 【打印】 【关闭】
6月22日,国际学术期刊 Briefings in Bioinformatics 以“Dr AFC: drug repositioning through anti-fibrosis characteristic”为题,在线发表了基于化合物抗纤维化特征的药物重定位平台Dr AFC(https://www.biosino.org/drafc/),为研究人员提供了准确、方便、快捷的在线药物重定位预测工具,助力临床新药研发。
药物重定位指将已有药物再利用,将其应用于新的疾病。与传统的药物研究相比,药物的重定位具有巨大的优势,其大大缩短药物研发周期,降低成本并降低潜在风险,现已成为药物研发中重要策略之一。然而,如何不断优化药物重定位方法和模型则成为目前需要攻克的难点。纤维化是许多疾病致病机制中的关键病理过程,这些涉及到纤维化的疾病具有共同的机制和治疗靶点,具有共同的治疗方法和药物。因此,研究团队开发了基于化合物抗纤维化特征的药物重定位预测算法,在预测模型中引入抗纤维化的特征,提高重定位的成功率。
该研究系统收集了纤维化相关的文献和抗纤维化临床实验数据,经过筛选和分析后,最终获得3096篇纤维化相关文献,1787个抗纤维化临床实验,1067个纤维化相关靶点,507个抗纤维化化合物-靶点的相互作用和1223个抗纤维化化合物,其中包括386个DrugBank中FDA批准的上市药物。基于此,该研究首先建立了抗纤维化药物知识库,进一步整合DrugBank上市药物小分子数据构建了“基于化合物结构谱预测模型”(Structure Profile Prediction Model, SPPM),和整合小分子刺激基因表达谱数据(CMap, connectivity map)构建了“基于化合物生物表达谱预测模型”(Biological Profile Prediction Model, BPPM)。在这两个药物重定位的模型中,根据化合物是否与纤维化有关来定义阳性和阴性数据,经过质控和预处理后用于后续模型的训练和测试。在评估4种不同的机器学习算法后,发现梯度提升算法具有最优的准确性和AUC,进而通过迭代特征消除(IFE, Iterative feature elimination)算法筛选并确定最优的特征(feature)组合,构建最终模型。模型在实际数据中进行了进一步测试与验证,得到了良好的预测效果。
该研究基于SPPM和BPPM模型开发了在线药物重定位平台Dr AFC,用户可以分别上传化合物的结构谱(通常为结构描述符/指纹)或生物表达谱(通常为化合物刺激的转录组表达值)来进行重定位分析。此外,Dr AFC平台中还加入了常用的药物设计分析功能,包括类药性分析、相似性计算、结构匹配等工具,方便研究人员使用。
该工作由同济大学吴顶峰、高文星、黎晓逸等在同济大学朱瑞新教授、中国科学院上海营养与健康研究所生物医学大数据中心张国庆研究员和中山大学朱立新教授的共同指导下完成。研究得到了科技部国家重点研发计划、国家自然科学基金、上海市自然科学基金、中国科学院等多项基金的资助。
在线服务地址: https://www.biosino.org/drafc/
文章链接:https://academic.oup.com/bib/article/doi/10.1093/bib/bbaa115/5860688
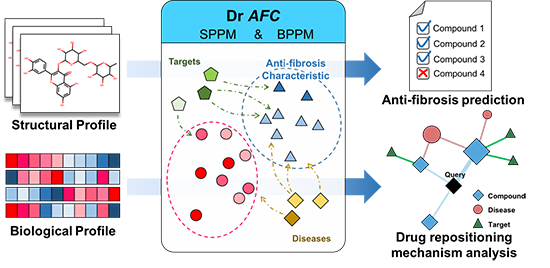
图:Dr AFC药物重定位平台构架。平台实现了基于化合物结构谱(SPPM)和基于化合物生物表达谱(BPPM)的药物重定位预测模型,集成了药物重定位预测和重定位机制分析等多种方便的新药研发工具。
